Presentación del Proyecto Final Química Ort 2008
Proyecto Química ORT del Genoma Humano
(Notese que el blog comienza abajo de todo, y cada nuevo dia se va poniendo encima del otro, quedando el final arriba)
Por cuarto año consecutivo los alumnos del último año de la especialidad Química de la Escuela Técnica ORT Argentina realizarán sus proyectos finales en distintas universidades y centros de investigación con profesionales reconocidos. La metodología de trabajo evita la transpocisión didáctica permitiendo a los estudiantes realizar su actividad final en los lugares donde se está generando el conocimiento.
La articulación entre el nivel medio y el postgrado universitario es auspiciada por el CONICET (Consejo Nacional de Investigaciones Científicas y Técnicas). Los 15 proyectos de investigación (incluyendo este) para el año en curso son:
Nuestro Proyecto: Estudio de la Growth Associated Protein (GAP-43), su interacción con la Ubicutina y su participación en el control del ciclo celular en células NIH3T3 transfectadas en forma estable y transiente. Efecto de la Apo-transferrina en la remielización: participación de la vía Notch en la diferenciación oligodendrioglial.
Investigador: Doctora Ana María Adamo
Instituto: Departamento de Química Biológica Patológica, Facultad de Farmacia y Bioquímica de la UBA. CONICET.
Alumnos: Rodrigo C. Pampin y Robby Mattes
1) Proyecto: Evaluación de la expresión de la molecula de adhesión cadherina epitelial en tejidos humanos normales y tumorales.
Investigador: Dra. Mónica Vazquez-Levin.
Instituto: Instituto de Biología y Medicina Molecular (IBYME - CONICET).
Alumnos: Yael Dobzewicz Y Gala Szapiro.
Link: http://www.proyecto-6q.blogspot.com/
2) Proyecto: Acción del hexaclorobenceno (HCB) sobre la uroporfirinogeno de una línea celular hepatocitos humanos. Mecanismo de acción.
Investigador: Dra. María del Carmen Ríos de Molina. Instituto: Departamento Química Biológica, Facultad de Ciencias Exactas y Naturales.
Alumnos: Lucas Toiw y Uriel Frid.
Link: http://www.proyectofrid-toiw08.blogspot.com/
3) Proyecto: Modelos experimentales de enfermedades metabólicas: Porfiria y Síndrome Metabólico. Proteómica y Metabolómica de estos disturbios.
Investigador: Dra. Marta Blanca Mazzetti.
Instituto: Departamento de Química Biológica, Facultad de Ciencias Exactas y Naturales (UBA).
Alumnos: Maria Duperron y Mauro Elencwajg.
Link: http://proyectofinal6q.blogspot.com/
4) Proyecto: Factores no hemodinámicos relacionados con la génesis y evolución de la proteinuria durante la enfermedad renal progresiva.
Investigador: Dra. Elsa Zotta.
Instituto: Laboratorio de Fisiopatogénia, Departamento de fisiología, Facultad de medicina, UBA.
Alumnos: Barbara Helueni y Melanie Naiman.
Link: http://www.proyectoq08.blogpsot.com/
5) Proyecto: Estudio de la participacion de la anandamida en la regulacion de la interaccion espermatozoide-ovioducto en un modelo bovino.
Investigador: Dra. Silvina Perez Martinez.
Instituto: Facultad de medicina - UBA
Alumnos: Judith Arenas Tenenbaum y Melina Braverman
Link: http://www.scienceproject08.blogspot.com/
6) Proyecto: El estudio de los mecanismos moleculares involucrados en la regulación del gen UGA4 de Saccharomyces cervisiae en respuesta a cambios en la disponibilidad de nutrientes con el fin de dilucidar las distintas cascadas de señales desencadenadas por dichos nutrientes y establecer sus interconexiones.
Investigador: Dra. Susana Correa García.
Instituto: Departamento de Química Biologica, Facultad de ciencias Exactas y Naturales, Universidad de Buenos Aires.
Alumnos: Iván Mikiej y Victoria Salama
Link: http://www.proyectoquimica22.blogspot.com/
7) Proyecto: Diagnóstico de la enfermedad de Von Willebrand (VWD) tipo 2N por técnicas fenotípicas y genotípicas.
Investigador: Dra. Adriana I. Woods.
Instituto: Instituto de Investigaciones Hematológicas "Mariano R. Castex" de la Academia Nacional de Medicina.
Alumnos: Abigail Skverer y Daniela Lin
Link: http://www.vwd-diagnostico.blogspot.com/
8) Proyecto: Estudio de la mielinogenesis en el sistema nervioso periferico en condiciones fisiologicas y patologicas. Participacion de celulas pluripotentes en el proceso de degeneracion-regeneracion nerviosa.
Investigador: Dra Patricia Setton-Avruj.
Instituto: Dpto de Quimica Biologica, Facultad de Farmacia y Bioquimica (IQUIFIB-UBA-CONICET).
Alumnos: Daniel Averbuj y Eitan Rozenszajn.
Link: http://www.proyectoaverbuj-rozenszajn.blogspot.com/
9) Proyecto: Diseño y desarrollo de vacunas antitumorales empleando bacterias y celulas tumorales modificadas con genes inmunomodiladores. Estudio de los mecanismos inmunes inducidos.
Investigador: Claudia I. Waldner y Claudia Mongini.
Instituto: Laboratorio de inmunologia celular y molecular. Centro de Estudios Farmacologicos y Botanicos (CEFYBO. CONICET-UBA)
Alumnos: Amalia Surijon y Maria Belen Tolava Rivero
Link: http://www.amibeluproyecto.blogspot.com/
10) Proyecto: Marcadores Geneticos Asociados al Cáncer
Investigador: Dr.Javier Hernán Cotignola
Instituto: Laboratorio de Cáncer y Apoptosis del Departamento de Quimica Biologica de la Facultad de Ciencias Exactas y Naturales de la Universidad de Buenos Aires.
Alumnos: Ayelén Marano y Solange Perchik
Link: http://marcadoresgeneticos.blogspot.com/
11) Proyecto: Expresion del canal de la conductancia transmembranal de la Fibrosis Quistica (CFTR) en placentas Preeclampticas: posible rol en la regulacion de la actividad de la Acuoporina-9 (AQP9).
Investigador: Dr Alicia E Damiano
Instituto: Catedra de Biologia Celular, Departamento de Ciencias Biologicas, Facultad de Farmacia y Bioquimica (UBA)
Alumnos: Gabriel Colodenco y Martín Sapir.
Link: http://www.tarkuselp.blogspot.com/
12) Proyecto: Proteómica de factores secretados por células de cáncer de mama y mama normal con capacidad inhibitoria de la producción lipídica.
Investigador: Dra.Guerra de Grignoli Liliana Noemi
Instituto: Departamento de Ciencias biologicas. Facultad de ciencias exactas y naturales,UBA-CONICET.
Alumnos: Fabiana Durante y Tamara Broitman.
Link: http://tyf-proyecto08.blogspot.com/
13) Proyecto: Neovascularizaciòn en un modelo murino de inflamaciòn aguda inducida por LPS.
Investigador: Eulalia de la Torre.
Instituto: Facultad de Medicina - Uba - Laboratorio de Inmunofarmacologia
Alumnos: Federico Mauas Walach, Pablo Kuleff.
Link: http://finalproyectort.blogspot.com/
14) Proyecto: Interacción de sumo-1 y mage-a2 en la regulacion del oncosupresor p53.
Investigador: -
Instituto: -
Alumnos: Yair Litman y Leonel Sterman
Link: http://proyectosumo.blogspot.com/
martes, 25 de noviembre de 2008
Presentacón
Presentación del Proyecto Final Química Ort 2008
Día 17: Conclusión Final
Los resultados presentados en esta monografía demuestran que se cumplieron los objetivos propuesto. La visualización de los conjugados de ubicuitina es una tarea dificultosa y hasta el momento las técnicas generalmente utilizadas para la detección de los mismos incluyen inmunoprecipitación seguida de Western Blot utilizando anticuerpos específicos. Esta técnica tiene limitaciones ya que no permite el análisis de la ubicuitinización en células in vivo.
La técnica de fluorescencia por complementación desarrollada por Deyu Fang and Tom K. Kerppola es una herramienta útil para la visualización de proteínas ubicuitinizadas en células in vivo. Una vez puesto a punto este sistema, el mismo podrá ser utilizado para la localización de conjugados de ubicuitina de cualquier proteína de interés. Teniendo en cuenta los antecedentes del grupo de trabajo, este sistema podrá ser utilizado para caracterizar la conjugación de GAP-43 a la ubicuitina.
Además, cabe destacar que en esta monografía se utilizaron técnicas de biología molecular siendo esta una herramienta muy útil para el estudio y compresión de diversos procesos celulares.
Día 16: Resultados de la tranfección
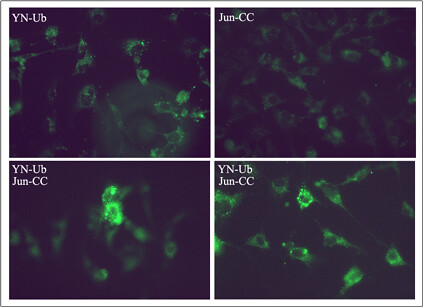
Se pudo obtener una visualización directa de los conjugados de Jun con ubicuitina en las células transfectadas con las distintas construcciones. Como se puede observar en la imagen de la figura 4, las células transfectadas con ambos plásmidos son fluorescentes y sorprendentemente esta fluorescencia se observa principalmente en pequeñas estructuras citoplasmáticas, visualizadas como un puntillado. La localización de estas estructuras es perinuclear. Por otro lado, las células transfectadas con los plásmidos solos no muestran una fluorescencia marcada y la distribución de la misma es diferente a la de las células cotransfectadas.
Este resultado confirma la factibilidad de la metodología, la identificación de conjugados de Jun con ubicuitina en células “in vivo” y sugiere que la ubicuitinización de Jun causa un transporte de esta proteína nuclear hacia estructuras citoplasmáticas como fue descripto por Deyu y colaboradores.
Día 15: Transfección
El día anterior al experimento se sembraron 3000 células en placa de 24 pocillos conteniendo cubre objetos de vidrio previamente tratados con poly-L lisina. En el momento de la transfección, las células estuvieron en un 70-80 % de confluencia, fase exponencial del crecimiento de las mismas donde el ingreso del plásmido esta favorecido. Para cada pocillo se obtuvo una mezcla de transfección que constó en la preparación en forma separada de dos soluciones. En una de ellas se diluyó 1 mg de DNA (ya sea el plásmido YN-Ub o el Jun-CC) en 200 ml de medio libre de suero y antibiótico. La otra solución contenía 5ml de LipofectaminaTM 2000 diluida en 200 ml de medio libre de suero y antibiótico.

Ambas soluciones se dejaron reposar aproximadamente 15 minutos. Luego de la incubación se combina el ADN diluido con la solución de LipofectaminaTM 2000 para la formación de los complejos y se le adicionan 600 ml del medio sin suero y sin antibiótico (volumen final 1 ml). Se mezcló suavemente y se incubó durante 45 minutos a temperatura ambiente. Mientras tanto la monocapa de células se lavó con medio libre de suero y antibiótico y cumplido los 45 minutos se le agregó a cada pocillo los complejos.

Las células se incubaron en estufa en su forma habitual y 5 horas más tarde se les agregó a cada pocillo 1ml de medio conteniendo 20% de suero fetal bovino, penicilina 100 U/ml y estreptom
icina 100 mg/ml (2 x el medio completo). De esta manera, al entrar en contacto con el medio presente, este se diluyó a su concentración habitual. Al día siguiente se cambia el medio de cultivo y se incuba hasta cumplidas las 24 hrs. post-transfección. Posteriormente se incubaron las células 6 hrs. a 30ºC para poder observar la complementación de fluorescencia ya que esto favorece la maduración del fluoróforo. Una vez cumplido ese tiempo las células fueron fijadas con paraformaldehido 4% en PBS durante 20 minutos. Luego se procedió a lavados exhaustivos con PBS para eliminar restos de paraformaldehido y finalmente los cubreobjetos de vidrio fueron montados en portaobjetos con medio de montaje para fluorescencia (Dako). Las células se mantienen en oscuridad. Se observo la fluorescencia de las células transfectadas con microscopio de fluorescencia (Olympus Optical CO, LTD, Japón) y se capturó la imagen de las mismas ya que este posee una cámara acoplada Olympus BX50.

Día 14: Cuantificación del ADN
La cantidad total de ADN presente en las muestras de los plásmidos purificados se determinó midiendo la absorbancia de una alícuota de dichos plásmidos a 260 nm y a 280 nm con un espectrofotómetro. La absorbancia a 260 nm se empleó para calcular la concentración de ADN. Una solución de 50 μg/ml proporciona una absorbancia de 1, por lo que la concentración de ADN en la muestra en μg/ml es igual a 50 x A260 nm. El cociente de absorbancias 260/280 se utiliza para calcular la pureza de la preparación. Un cociente inferior a 1.8 indica que hay impureza.
| | 260nm | 280nm | 260/280 | Concentración |
| YN-Ub | 0,344 | 0,227 | 1.5 | 0.592 μg/μl |
| Jun-CC | 0,932 | 0,605 | 1.5 | 2.33 μg/μl |
Día 13: Midiprep
Se realizó una midiprep para la purificación del ADN utilizando un kit comercial de Wizard Plus Midipreps DNA Purification System (Promega) que permite un método de aislamiento rápido y confiable para el ADN. Una vez obtenido el ADN, se realizó una nueva digestión enzimática para realizar una electroforesis en geles de azarosa 1% para la cuantificación utilizando un estándar גHind III.
Aqui se puede observar el procedimiento de la midiprep.

jueves, 2 de octubre de 2008
Día 12: Finalizamos el Western
Día 11: Empezamos un Western
Empezamos armando el gel de poliacrilamida en presencia de SDS, una diferencia con el Northren-blot es que, a diferencia del Northren que es horizontal, esta corrida se hace en vertical.
Al finalizar la corrida, se coloca una membrana PVDF encima del gel, y se le hace pasar corriente eléctrica al igual que en la corrida, solo que esta vez las muestras, en vez de avanzar por las calles, pasan del gel a la membrana.
Día 10: Conceptos
Día 8: Sala de Cultivo
jueves, 14 de agosto de 2008
Día 7: Electroforesis
Se evaluó la presencia de los plásmidos mediante una electroforesis en un gel de agarosa 1% (0,5gr de agarosa, 50ml de buffer TBE [2,7gr Tris, 1,3075 de ácido bórico, 1ml de EDTA 0,5 molar pH 8]) y 4μl de Bromuro de Etidio.
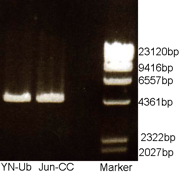
Día 6: Obtención del plásmido para su control II
Posteriormente, tomamos 1,5 ml de cada uno de los cultivos de las bacterias que crecieron correctamente (YN-Ub) y la centrifugamos a 10000 x g durante 2 minutos.
Preparamos una mezcla de buffer Telt conteniendo Lisozima, usamos 10 ul Lisozima y 90 ul de telt por tubo, y se la agregamos a las suspensiones de bacterias, luego agitamos utilizando un vortex para lograr una buena resuspensión.
Luego seguimos el siguiente procedimiento:
Dejamos las muestras 5 minutos a temperatura ambiente.
Hervimos las muestras durante 1 minuto.
Las dejamos en hielo por 2 minutos.
Centrifugamos 15 minutos a 10000 x g.
Recogimos el sobrenadante.
Agregamos 1 volumen equivalente de isopropanol.
Las dejamos 20 minutos a temperatura ambiente.
Centrifugamos durante 20 minutos a 10000 x g y descartamos el sobrenadante.
Agregamos 10 ul de EtOH 70% (v/v).
Centrifugamos 5 minutos a 10000 x g.
En ese momento nos teníamos que ir y Karina terminó de secar las muestras al vacío y las resuspendió en 10 ul de agua estéril.
Se conservaron las muestras a -20ºC hasta la semana siguiente.
Día 5: Obtención del plásmido para su control I
Se observaron colonias en las placas con las bacterias transformadas con el plásmido YN-Ub pero solo unas pocas colonias dispersas se observaron en las placas con las bacterias transformadas con el plásmido Jun-CFP.
Seleccionamos 2 colonias aisladas de cada placa, las colocamos en el medio de cultivo LB con el ATB correspondiente en cada caso y las dejamos incubando over-night a 37ºC.
jueves, 26 de junio de 2008
Dia 4: Transformación de Bacterias
Para cada transformación, a 100 ul de la suspensión de bacterias competentes se le agrega 1 ug de ADN (plásmido) en un tubo eppendorf y se siguió el siguiente protocolo:
- Incubar 30 minutos a 4ºC (en hielo).
- Incubar 1 minuto a 42ºC.
- Se llevó nuevamente la preparación 2 minutos a 4ºC (en hielo).
- Finalmente se agregaron 300 ul de LB, sin antibiótico, y se incubó durante 1 hora a 37 ºC con agitación.
- Por último se sembraron 50 ul de la suspensión de bacterias transformadas en placas de LB/agar en presencia de los antibióticos de selección según corresponde a cada plásmido.


Las placas se incubaron durante aproximadamente 12 hs a 37ºC y solo las bacterias que hallan incorporado el plásmido, y por lo tanto resistentes al antibiótico, formarán colonias.
miércoles, 4 de junio de 2008
Día 3:Práctica de Electroforosis

Esperamos a que se seque el gel y lo preparamos para la electroforesis, colocando además la solución con la que hicimos el gel para mantener el mismo ph. Una vez realizado esto, sembramos.

En la primera calle se encuentra el patrón, en la tercera, la primera muestra sembrada por nosotros, se puede ver que quedo un poco afuera, ya que como las calles son chicas, con falta de experiencia basta para errarle, y por eso mismo estamos practicando. Mientras dejábamos que corra, sacamos los instrumentos que habíamos dejado en la autoclave y preparamos las placas de petri. La corrida es bastante parecida a una cromatografía. Nosotros sembramos plásmidos (sin cortar), y estos tienen colorante violeta, que va corriendo "delante" de nuestras muestras.

En la imagen se puede ver muy bien el colorante de nuestras muestras, el amarillo del patrón, y apenas, el violeta y turquesa del patrón (abajo hay un link a fotos mas grandes). Nuestra muestra debería estar al nivel del turquesa, pero hasta no "revelar" el gel, no hay forma de saberlo. Para verlo hay dos formas (aunque sea, usamos dos formas), una es directamente con luz ultravioleta, y la otro con un aparato que te da la imagen en la computadora. El resultado fue el siguiente:
 Por alguna razón, la primera muestra no se revelo, sin embargo la segunda si. Se puede ver que la mayor parte coincide con una parte del patrón (a la altura de el colorante turquesa), la razón por la que salio tan corrida la muestra es que no cortamos el plásmido, y por lo tanto este es redondo, creando ciertas complicaciones en la corrida.
Por alguna razón, la primera muestra no se revelo, sin embargo la segunda si. Se puede ver que la mayor parte coincide con una parte del patrón (a la altura de el colorante turquesa), la razón por la que salio tan corrida la muestra es que no cortamos el plásmido, y por lo tanto este es redondo, creando ciertas complicaciones en la corrida.
jueves, 22 de mayo de 2008
Día 2: Inicio

Posteriormente hicimos una visita al lugar, nos mostraron las distintas instalaciones, el cuarto de cultivo y finalmente preparamos todo el material para esterilizar para comenzar con el trabajo la semana que viene.
Ah! Además vimos como Verónica, que trabaja en el otro proyecto mencionado arriba, inyectaba una proteína, la apotransferrina en cerebro de las ratas. Ella usa un equipo especial que se llama estereotáxico y permite inyectar distintas drogas o sustancias en áreas específicas del cerebro de rata estableciendo previamente las coordenadas. Nos dijeron que además de seguir con nuestro proyecto nos iban a mostrar distintas técnicas que se usan en el laboratorio en relación a los dos proyectos mencionados
Esto es lo que estaremos haciendo durante el año; el proceso es el siguiente:
Día 1: Presentación
martes, 22 de abril de 2008
Bienvenidos
Este blog será usado por los alumnos del colegio técnico ORT, Rodrigo Cesáreo Pampin y Robby Mattes para mostrar las distintas experiencias que irán adquiriendo durante su pasantía en un laboratorio donde se llevan a cabo tareas de investigación.
Esta pasantía se desarrollará en la cátedra de Química Biológica Patológica del Departamento de Química Biológica, de la Facultad de Farmacia y Bioquímica de la Universidad de Buenos Aires. El grupo de investigación, con el cuál trabajaremos está liderado por la Dra. Ana M. Adamo y con ella trabajan, Karina De Moliner, Verónica Millanovich y Evangelina Aparicio, las tres estudiantes de Doctorado y Berenice Milanesio, estudiante de grado de Bioquímica.
En este laboratorio se están llevando a cabo dos proyectos de investigación:
1) Título: La Growth Associated Protein (GAP-43), su interacción con la Ubicuitina y su participación en el control del ciclo celular en células NIH3T3 transfectadas en forma estable y transiente.
Fuente de financiamiento: Universidad de Buenos Aires, Secretaría de Ciencia y Técnica, Programación Científica 2004-2007 (Subsidio B032). Directora
2) Titulo: “Efecto de la apo-transferrina en la remielinización: participación de la vía de señalización mediada por el receptor Notch en la diferenciación oligodendroglial
Fuente de financiamiento: Subsidio de la Agencia Nacional de Promoción Científica y Tecnológica. PICT 2004 Nº 25408.
Nuestro trabajo, durante la pasantía, consistirá en realizar trabajos de apoyo a la investigación en el marco del proyecto, y en particular trabajaremos con Karina De Moliner y con Berenice Milanesio para poner a punto un método que permitirá evaluar la conjugación de GAP-43 a Ubicuitina “in vivo” en las células en cultivo mediante un estudio de complementación de fluorescencia. Durante este trabajo haremos tranformaciones en bacterias utilizando distintos plásmidos, geles de agarosa para evaluar su presencia en las bacterias, purificación de los mismos y transfección en células eucariontes. En este blog iremos volcando todas nuestras experiencias con los resultados obtenidos.